Classification
L'analyse par biopuces d'un tissu animal ou végétal fournit une photographie de l'état du transcriptome au sein de ce tissu. Cette photographie peut être utilisée pour caractériser le tissu, et pour comparer différents tissus ou différentes conditions. Par exemple, on peut espérer détecter des «signatures» particulières au sein du transcriptome correspondant à diverses pathologies (telles que les cancers) ou à divers états physiologiques d'une plante. Caractériser la signature d'une pathologie au niveau du transcriptome fournit un moyen de diagnostiquer cette pathologie avant que des signes physiologiques n'apparaissent, et ainsi de la traiter rapidement et donc plus efficacement et à moindre coût. Dans le monde végétal, cette approche peut permettre de détecter et caractériser les gènes qui interviennent dans la réponse d'une plante à une carence nutritionelle, par exemple en phosphate.
D'un point de vue informatique, la caractérisation de ces signatures se traduit naturellement en problèmes de classification, supervisés et non-supervisés. Quelle que soit la problématique, une des difficultés majeures à laquelle les algorithmes sont confrontés est la qualité souvent médiocre des données : bruit élevé, grande variabilité, données manquantes, classes recouvrantes, etc. C'est pourquoi les approches de types probabilistes et statistiques semblent particulièrement bien adaptées.
Cela peut être le diagnostic de maladies (eg. malade ou sain), la prédiction de l'issue d'une maladie (guérison vs. mort), la recherche de l'état d'une plante (carencée vs. non carencée), etc. Ce problème est un défi pour les algorithmes d'apprentissage classiques qui ont été étudiés pour travailler avec beaucoup d'exemples et peu de variables. Ici au contraire, les variables (gènes) se content par milliers, alors que les exemples (tissus) sont généralement de l'ordre de quelques dizaines.
Les algorithmes les plus utilisés dans le contexte des biopuces sont :
- Méthodes probabilistes
- Les grands classiques linéaires et quadratiques sont passés en revue dans [Dudoit et al., 2002].
- Weighted Voting
- Un classifieur par votes pondérés introduit dans [Golub et al., 1999].
- KNN
- On peut voir par exemple [Dudoit et al., 2002].
- Arbres de décision
- Voir [Dudoit et al., 2002].
- Support Vector Machine
- On peut voir [Brown et al., 2000].
Ici sont listés quelques jeux de données pouvant servir au test de méthodes de classification supervisées, ainsi que les références d'un ou plusieurs papiers ayant déjà utilisé ces données dans ce contexte.
- Leukemia 1
- [Dudoit et al., 2002] comparent KNN, CART, diverses approches paramétriques probabilistes (e.g. Naives Bayes classifier), ainsi que l'ajout de Bagging et Boosting. [Ben-Dor et al., 2000] comparent KNN, SVM et Boosting.
- Lymphoma 1
- [Dudoit et al., 2002] comparent KNN, CART, diverses approches paramétriques probabilistes (e.g. Naives Bayes classifier), ainsi que l'ajout de Bagging et Boosting, sur un sous-ensemble des données limité à 3 classes.
- Colon cancer
- [Ben-Dor et al., 2000] comparent KNN, SVM et Boosting.
- Lymphoma 2
- [Shipp et al., 2002] comparent KNN, Weighted Voting, et SVM.
- NCI 60
- [Dudoit et al., 2002] comparent KNN, CART, diverses approches paramétriques probabilistes (e.g. Naives Bayes classifier), ainsi que l'ajout de Bagging et Boosting sur un sous-ensemble limité à 8 classes.
- Breast cancer
- [van 't Veer et al., 2002] rapportent les résultats de leur méthode de classification (construction d'un profil moyen pour chaque classe et recherche du profil moyen le plus proche pour classer un nouvel exemple).
- Brain cancer
- [Pomeroy et al., 2002] rapportent les résultats obtenus par KNNs, Weighted Voting, SVMs, etc.
On distingue deux type de problème pour la classification non supervisée, suivant que l'on veuille classer les tissus ou les gènes :
- Pour le clustering de tissus, cela peut être par exemple l'identification d'éventuels sous-types d'une certaine forme de cancer. Ce type de problème souffre des mêmes verrous que pour la classification supervisée, c'est-à-dire peu d'exemples et beaucoup de variables.
- Pour le clustering de gènes, on cherche à caractériser des groupes de gènes qui sont co-régulés, c'est-à-dire qui "réagissent" simultanéments : leur niveau d'expression augmente ou diminue dans les mêmes conditions, dans les même temps, etc.
Les algorithmes les plus utilisés dans le contexte des biopuces sont :
- Hierarchical clustering :
- Logiciels CLUSTER et TREEVIEW [Eisen et al., 1998] disponible sur http://rana.lbl.gov/EisenSoftware.htm.
- SOM :
- Introduit par [Kohonen, 1997]. Logiciel GENECLUSTER [Tamayo et al., 1999] disponible sur http://www-genome.wi.mit.edu/cancer/software/software.html.
- CLICK :
- Introduit par [Sharan and Shamir, 2000]. Logiciel disponible sur http://www.cs.tau.ac.il/~rshamir/expander/expander.html.
- CAST :
- Introduit par [Ben-Dor et al., 1999]. Logiciel BIOCLUST. Disponible ?
- K-MEANS :
- Une version du k-means [Herwig et al., 1999]. Disponible ?
Ici sont répertoriés quelques jeux de données pouvant servir au test de méthodes de classification non supervisées. Pour chaque jeu de données sont mentionnés des clusters biologiques potentiels qui peuvent servir à évaluer les résultats obtenus. Sont également référencés un ou plusieurs papiers ayant déjà utilisé ces données dans ce contexte.
- Colon Cancer
- Évaluation biologique des clusters par la séparation tumeur vs. normal. [Sharan et al., 2002] comparent résultats CLICK et CAST.
- Leukemia 1
- Évaluation biologique des clusters par la séparation AML vs. ALL. [Sharan et al., 2002] comparent résultats CLICK et CAST. [Golub et al., 1999] utilisent SOM. Également utilisé par [Ben-Dor et al., 2001].
- Leukemia 2
- Évaluation biologique des clusters par la séparation B-cell vs. T-cell. [Sharan et al., 2002] comparent les résultats obtenus par CLICK et CAST, ainsi que l'effet d'une procédure de sélection des 50 meilleurs gènes.
- Lymphoma 1
- Évaluation biologique des clusters par la séparation des classes originales. [Alizadeh et al., 2000] utilisent le clustering hiérarchique. Données également utilisées dans [Ben-Dor et al., 2001].
- Yeast Cell-Cycle 1
- Clusters biologiques potentiels : les gènes mis en jeu lors des différentes phases du cycle cellulaire : M, G1, S, et G2. [Tamayo et al., 1999] utilisent la méthode SOM. [Sharan et al., 2002] utilisent la méthode CLICK.
- Yeast Cell-Cycle 2
- Même clusters biologiques potentiels que Yeast Cell-Cycle 1. Comparaison des méthodes SOM, CLICK, CAST, K-Means sur un sous-ensemble des données dans [Sharan et al., 2002].
- Yeast Regulatory motif
- Évaluation biologique des clusters par recherche des motifs de régulation communs aux gènes d'un même cluster via le logiciel ALIGNACE http://atlas.med.harvard.edu/. [Sharan et al., 2002] comparent les résultats obtenus par CLICK et SOM.
- Brain Cancer
- [Pomeroy et al., 2002] utilisent SOM et Hierachical Clustering.
Ici sont référencés quelques jeux de données de biopuces du domaine public, plus ou moins classiques, et (à deux exceptions près) normalisées. Sont indiqués après le nom des données, le nom de l'organisme biologique en question, la référence du papier dans lesquelles elles ont été publiées, une courte présentation de leur contenu et l'URL d'où elles peuvent être téléchargées.
- Yeast Cell-Cycle 1
- Saccharomyces cerevisiae. [Cho et al., 1998]. 6218 gènes. 14 mesures/gènes effectuées lors de 2 cycles cellulaires à 10 minutes d'intervalles pendant 160 minutes. Données Affymetrix normalisées : http://genomics.stanford.edu/.
- Yeast Cell-Cycle 2
- Saccharomyces cerevisiae. [Spellman et al., 1998]. 6178 gènes dans 79 conditions différentes. Données puces cDNA non normalisées : http://genome-www.stanford.edu/cellcycle/.
- Yeast Regulatory motif
- Saccharomyces cerevisiae. [Jelinsky et al., 2000]. 6200 gènes dans 26 conditions différentes. Données Affymetrix normalisées : http://www.hsph.harvard.edu/geneexpression/index1.htm.
- Colon Cancer
- Homo sapiens. [Alon et al., 1999]. 2000 gènes mesurés sur 62 tissus (40 tumeurs et 22 "normaux"). Données Affymetrix non normalisées : http://microarray.princeton.edu/oncology/affydata/index.html.
- Leukemia 1
- Homo sapiens. [Golub et al., 1999].
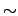 7000 gènes sur 72 patients (25 de type AML et 47 de type ALL). Données Affymetrix normalisées : http://www-genome.wi.mit.edu/cgi-bin/cancer/publications/pub_paper.cgi?mode=view&paper_id=43.
7000 gènes sur 72 patients (25 de type AML et 47 de type ALL). Données Affymetrix normalisées : http://www-genome.wi.mit.edu/cgi-bin/cancer/publications/pub_paper.cgi?mode=view&paper_id=43.
- Leukemia 2
- Homo sapiens. Données de Leukemia 1 restreint aux 47 patients ALL qui se divisent en 2 autres sous catégories (B-cell ou T-cell).
- Lymphoma 1
- Homo sapiens. [Alizadeh et al., 2000].
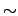 4000 gènes sur 96 patients (46 de type DLBCL et 50 appartenant à 8 autres classes). Données puces cDNA normalisées : http://llmpp.nih.gov/lymphoma/data.shtml (Figure 1).
4000 gènes sur 96 patients (46 de type DLBCL et 50 appartenant à 8 autres classes). Données puces cDNA normalisées : http://llmpp.nih.gov/lymphoma/data.shtml (Figure 1).
- Lymphoma 2
- Homo sapiens. [Shipp et al., 2002]. 7129 gènes sur 58 patients (32 "guéris" et 26 morts ou rechutes). Données Affymetrix normalisées : http://www-genome.wi.mit.edu/cgi-bin/cancer/publications/pub_paper.cgi?mode=view&paper_id=66.
- NCI 60
- Homo sapiens. [Ross et al., 2000]. 9703 gènes sur 60 tissus cancéreux (7 breast, 5 central nervous system, 7 colon, 6 leukemia, 8 melanoma, 9 NSCLC, 6 ovarian, 2 prostate, 9 renal, 1 inconnu). Données puces cDNA normalisées http://genome-www.stanford.edu/nci60/.
- Breast cancer
- Homo sapiens. [van 't Veer et al., 2002].
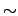 24000 gènes sur 78 patients (34 qui ont développés des métastases sous 5 ans et 44 qui n'en ont pas développées). Données puces Agilent normalisées http://www.rii.com/publications/2002/vantveer.htm.
24000 gènes sur 78 patients (34 qui ont développés des métastases sous 5 ans et 44 qui n'en ont pas développées). Données puces Agilent normalisées http://www.rii.com/publications/2002/vantveer.htm.
- Brain cancer
- Homo sapiens. [Pomeroy et al., 2002].
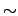 7000 gènes sur 60 patients (39 qui ont succombés et 21 qui sont restés en vie après le traitement). Données puces Affymetrix http://www-genome.wi.mit.edu/mpr/CNS/.
7000 gènes sur 60 patients (39 qui ont succombés et 21 qui sont restés en vie après le traitement). Données puces Affymetrix http://www-genome.wi.mit.edu/mpr/CNS/.
Quelques institutions proposent également une liste de pointeurs sur leur données (et d'autres). Ces données incluent celles déjà listées :
Enfin, il y a aussi des URLs qui pointent sur ces institutions, comme par exemple : http://linkage.rockefeller.edu/wli/microarray/data.html
- Alizadeh et al., 2000
-
Alizadeh, A. A., Eisen, M. B., Davis, R. E., Ma, C., Lossos, I. S., Rosenwald,
A., Boldrick, J. C., Sabet, H., Tran, T., Yu, X., Powell, J. I., Yang, L.,
Marti, G. E., Moore, T., Hudson, J. J., Lu, L., Lewis, D. B., Tibshirani, R.,
Sherlock, G., Chan, W. C., Greiner, T. C., Weisenburger, D. D., Armitage,
J. O., Warnke, R., and Staudt, L. M. (2000).
Distinct types of diffuse large B-cell lymphoma identified by gene
expression profiling.
Nature, 403(6769):503-511.
- Alon et al., 1999
-
Alon, U., Barkai, N., Notterman, D. A., Gish, K., Ybarra, S., Mack, D., and
Levine, A. J. (1999).
Broad patterns of gene expression revealed by clustering analysis of
tumor and normal colon tissues probed by oligonucleotide arrays.
Proc Natl Acad Sci USA, 96(12):6745-6750.
- Ben-Dor et al., 2000
-
Ben-Dor, A., Bruhn, L., Friedman, N., Nachman, I., Schummer, M., and Yakhini,
Z. (2000).
Tissue classification with gene expression profiles.
J Comput Biol, 7(3-4):559-583.
- Ben-Dor et al., 2001
-
Ben-Dor, A., Friedman, N., and Yakhini, Z. (2001).
Class discovery in gene expression data.
In RECOMB, pages 31-38.
- Ben-Dor et al., 1999
-
Ben-Dor, A., Shamir, R., and Yakhini, Z. (1999).
Clustering gene expression patterns.
J Comput Biol, 6(3-4):281-97.
- Brown et al., 2000
-
Brown, M. P. S., Grundy, W. N., Lin, D., Cristianini, N., Sugnet, C., Furey,
T. S., Manuel Ares, J., and Haussler, D. (2000).
Knowledge-based analysis of microarray gene expression data using
support vector machines.
Proceedings of the National Academy of Sciences, 1:262-267.
- Cho et al., 1998
-
Cho, R. J., Campbell, M. J., Winzeler, E. A., Steinmetz, L., Conway, A.,
Wodicka, L., Wolfsberg, T. G., Gabrielian, A. E., Landsman, D., Lockhart,
D. J., and Davis, R. W. (1998).
A genome-wide transcriptional analysis of the mitotic cell cycle.
Mol Cell, 2(1):65-73.
- Dudoit et al., 2002
-
Dudoit, S., Fridlyand, J., and Speed, T. P. (2002).
Comparison of discrimination methods for the classification of tumors
using gene expression data.
Journal of the American Statistical Association,
97(457):77-87.
- Eisen et al., 1998
-
Eisen, M. B., Spellman, P. T., Brown, P. O., and Botstein, D. (1998).
Cluster analysis and display of genome-wide expression patterns.
Proc Natl Acad Sci U S A, 95(25):14863-14868.
- Golub et al., 1999
-
Golub, T. R., Slonim, D. K., Tamayo, P., Huard, C., Gaasenbeek, M., Mesirov,
J. P., Coller, H., Loh, M. L., Downing, J. R., Caligiuri, M. A., Bloomfield,
C. D., and Lander, E. S. (1999).
Molecular classification of cancer: class discovery and class
prediction by gene expression monitoring.
Science, 286(5439):531-537.
- Herwig et al., 1999
-
Herwig, R., Poustka, A. J., Muller, C., Bull, C., Lehrach, H., and O'Brien, J.
(1999).
Large-scale clustering of cDNA-fingerprinting data.
Genome Res, 9(11):1093-105.
- Jelinsky et al., 2000
-
Jelinsky, S. A., Estep, P., Church, G. M., and Samson, L. D. (2000).
Regulatory networks revealed by transcriptional profiling of damaged
saccharomyces cerevisiae cells: Rpn4 links base excision repair with
proteasomes.
Mol Cell Biol, 20(21):8157-8167.
- Kohonen, 1997
-
Kohonen, T. (1997).
Self-Organizing Maps.
Springer.
- Pomeroy et al., 2002
-
Pomeroy, S. L., Tamayo, P., Gaasenbeek, M., Sturla, L. M., Angelo, M.,
McLaughlin, M. E., Kim, J. Y. H., Goumnerova, L. C., Black, P. M., Lau, C.,
Allen, J. C., Zagzag, D., Olson, J. M., Curran, T., Wetmore, C., Biegel,
J. A., Poggio, T., Mukherjee, S., Rifkin, R., Califano, A., Stolovitzky, G.,
Louis, D. N., Mesirov, J. P., Lander, E. S., and Golub, T. R. (2002).
Prediction of central nervous system embryonal tumour outcome based
on gene expression.
Nature, 415(6870):436-442.
- Ross et al., 2000
-
Ross, D. T., Scherf, U., Eisen, M. B., Perou, C. M., Rees, C., Spellman, P.,
Iyer, V., Jeffrey, S. S., de Rijn, M. V., Waltham, M., Pergamenschikov, A.,
Lee, J. C., Lashkari, D., Shalon, D., Myers, T. G., Weinstein, J. N.,
Botstein, D., and Brown, P. O. (2000).
Systematic variation in gene expression patterns in human cancer cell
lines.
Nat Genet, 24(3):227-235.
- Sharan et al., 2002
-
Sharan, R., Elkon, R., and Shamir, R. (2002).
Cluster analysis and its applications to gene expression data.
Ernst Schering Res Found Workshop, 2(38):83-108.
- Sharan and Shamir, 2000
-
Sharan, R. and Shamir, R. (2000).
Click: a clustering algorithm with applications to gene expression
analysis.
In Proc Int Conf Intell Syst Mol Biol, pages 307-16.
- Shipp et al., 2002
-
Shipp, M. A., Ross, K. N., Tamayo, P., Weng, A. P., Kutok, J. L., Aguiar, R.
C. T., Gaasenbeek, M., Angelo, M., Reich, M., Pinkus, G. S., Ray, T. S.,
Koval, M. A., Last, K. W., Norton, A., Lister, T. A., Mesirov, J., Neuberg,
D. S., Lander, E. S., Aster, J. C., and Golub, T. R. (2002).
Diffuse large B-cell lymphoma outcome prediction by gene-expression
profiling and supervised machine learning.
Nat Med, 8(1):68-74.
- Spellman et al., 1998
-
Spellman, P. T., Sherlock, G., Zhang, M. Q., Iyer, V. R., Anders, K., Eisen,
M. B., Brown, P. O., Botstein, D., and Futcher, B. (1998).
Comprehensive identification of cell cycle-regulated genes of the
yeast saccharomyces cerevisiae by microarray hybridization.
Mol Biol Cell, 9(12):3273-3297.
- Tamayo et al., 1999
-
Tamayo, P., Slonim, D., Mesirov, J., Zhu, Q., Kitareewan, S., Dmitrovsky, E.,
Lander, E. S., and Golub, T. R. (1999).
Interpreting patterns of gene expression with self-organizing maps:
methods and application to hematopoietic differentiation.
Proc Natl Acad Sci U S A, 96(6):2907-2912.
- van 't Veer et al., 2002
-
van 't Veer, L. J., Dai, H., van de Vijver, M. J., He, Y. D., Hart, A. A. M.,
Mao, M., Peterse, H. L., van der Kooy, K., Marton, M. J., Witteveen, A. T.,
Schreiber, G. J., Kerkhoven, R. M., Roberts, C., Linsley, P. S., Bernards,
R., and Friend, S. H. (2002).
Gene expression profiling predicts clinical outcome of breast cancer.
Nature, 415(6871):530-536.
This document was generated using the
LaTeX2HTML translator Version 2K.1beta (1.48)
Copyright © 1993, 1994, 1995, 1996,
Nikos Drakos,
Computer Based Learning Unit, University of Leeds.
Copyright © 1997, 1998, 1999,
Ross Moore,
Mathematics Department, Macquarie University, Sydney.
The command line arguments were:
latex2html -split 1 -link 1 -local_icons -no_navigation pointeurs.tex
The translation was initiated by on 2003-11-04
2003-11-04