Responsables :
Olivier Strauss, LIRMM, département robotique. 04 67 41 85 87. Noura Faraj, LIRMM, département informatique. 04 67 41 85 14.
Informations :
Réagissez sur le module image en envoyant un e-mel. Pour s'inscrire à ce module, remplissez ce formulaire formulaire.
Pensez par ailleurs à vous inscrire auprès de l'ADUM !
Contenu du module :
Ce module s'adresse aux étudiants de l'école doctorale I2S et aux chercheurs interessés par les techniques de traitement d'image et de vision par ordinateur. Les thèmes abordés dans ces conférences sont très larges. Ils portent généralement à la fois sur l'aspect fondamental du traitement d'image ainsi que sur l'aspect applicatif. Les conférenciers présentent un thème fort de leur équipe de recherche dont leur laboratoire est spécialiste au niveau national et international. Cliquez ici pour accéder au programme du module image des années précédentes.
Organisation du module :
Ce module de l'école doctorale est composé de 6 cours/conférences. Les conférences, d'une durée de 2 à 3 heures, se déroulent sur trois jours à raison d'une conférence par demi-journée. Les repas de midi sont pris en charge par nos partenaires pour permettre aux participants de discuter entre eux et avec les conférenciers.
Programme Synthétique
Lundi 26 mai
- 9 - 12h : Imagerie multidimensionnelle de polarisation : théorie et applications, Samia Ainouz Pr, INSA Rouen Normandie.
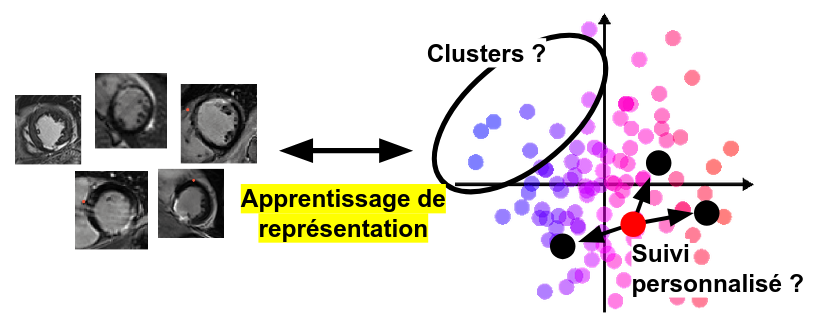
- 14 - 17h : Apprentissage de représentation et fusion d'informations pour l'analyse de populations en imagerie cardiaque, Nicolas Duchateau McF, Université Lyon 1, CREATIS.
Mardi 27 mai
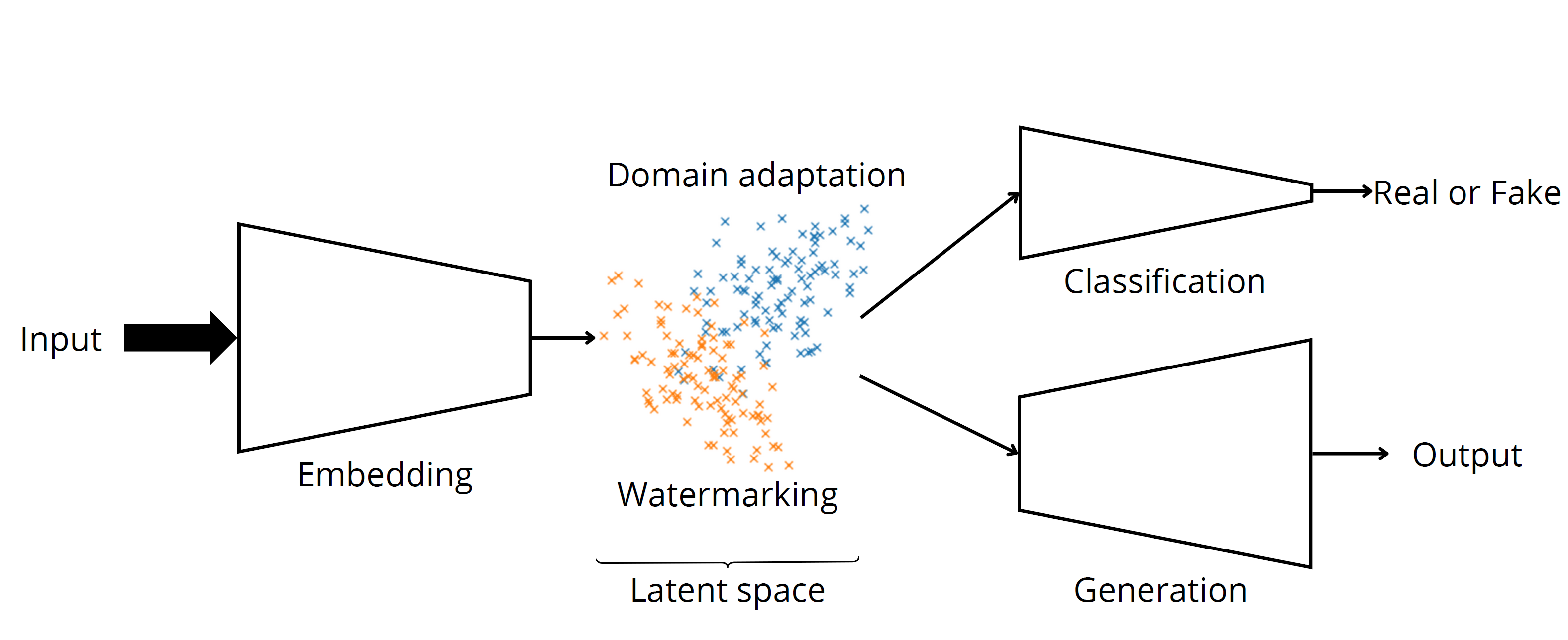
- 9 - 12h : Image Forensics & Security: Trends and Open Challenges, Vincent Itier McF, laboratoire CRIStAL Lille.
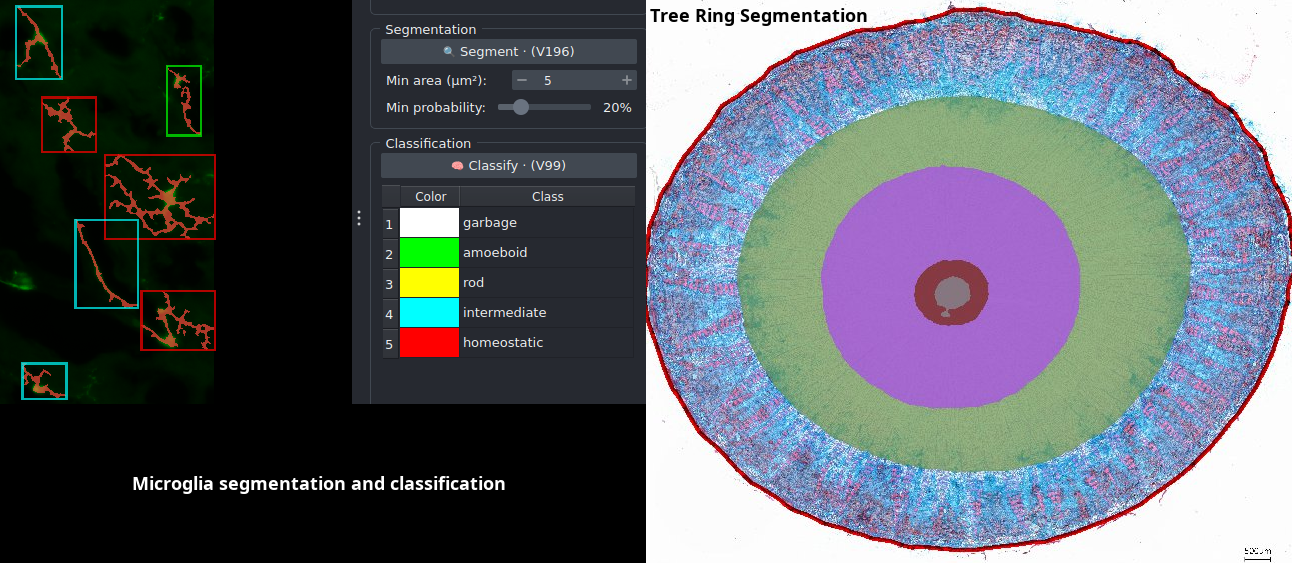
- 14 - 17h : Bioimage analysis: Introduction and application examples, Volker Baecker and Clément Benedetti Bioimage Analyst and Software Developers at INSERM.
Mercredi 28 mai
- 9 - 12h : Production virtuelle et environnements numériques chez Les Tontons Truqueurs Pierre-Marie Boyé et François Desrichard Directeur des Productions et Ingénieur R&D chez Les Tontons Truqueurs.
- 14 - 17h : Rendu expressif : approches orientées artistes et innovations en contrôle utilisateur David Vanderhaeghe McF, Université de Toulouse, l'IRIT.
Lieu
Les exposés du module image de l'école doctorale auront lieu dans l’amphithéâtre A36.02 du bâtiment 36 du campus Triolet de l’Université de Montpellier - Pl. Eugène Bataillon, 34090 Montpellier. Pour obtenir le plan, cliquez ici.