Les recherches publiées ici présentent un nouveau modèle probabiliste permettant d’estimer les vitesses de migration de pathogènes de manière bien plus précise que les techniques pre-existantes. Cette avancée a un impact en terme de santé publique et de prévention car ce nouveau modèle nous permet potentiellement de prédire les zones géographiques à fort risque d’être impactées par une épidémie.
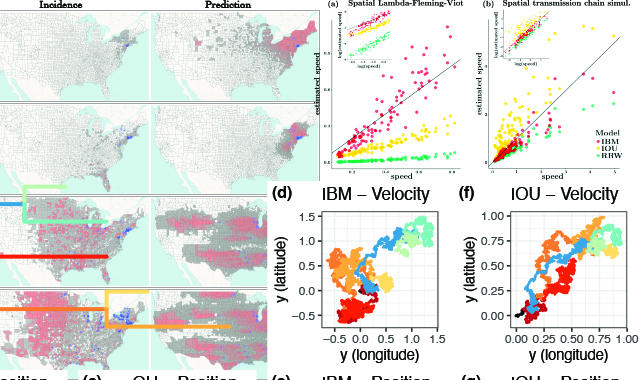
Un nouveau modèle permettant d’analyser conjointement des séquences génétiques ainsi que leurs coordonnées spatiales. L’étude des corrélations entre les distances physiques et génétiques entre séquences d’ADN nécessite la mise au point de techniques issues de l’approche phylogénétique. Cette dernière est couplée à la modélisation probabiliste des processus aléatoires qui caractérisent la diffusion spatiale. L’approche dite phylogéographique est utilisée notamment pour suivre l’évolution et la diffusion de virus lors d’épidémies.
Les modèles utilisés jusqu’ici ne permettaient cependant pas d’estimer correctement la vitesse de migration des pathogènes au cours de leur évolution. En collaboration avec l’IRD, l’université catholique de Louvain en Belgique et UCLA aux Etats-Unis, nous avons proposé un nouveau modèle de diffusion spatiale. Nos résultats montrent que celui-ci permet d’estimer la vitesse de diffusion de manière plus précise que les approches classiques. Fort de ces résultats, ce modèle a été appliqué avec succès à la prédiction de la diffusion du West Nile Virus (WNV) aux Etats-Unis: à partir de l’analyse des séquences virales et de leurs coordonnées spatiales, notre modèle est capable de prédire les régions probables de circulation des souches virales sur un horizon temporel d’une année. Il s’agit ici d’une des premières application où une méthode permettant de reconstruire l’évolution passée est mise à profit pour anticiper le futur.
Contact : stephane.guindon@lirmm.fr